Nat.Mach.Intell.| 基于双重众包的RNA降解预测模型
本文介绍一篇来自斯坦福大学的研究团队最近发表在Nature Machine intelligence期刊上名为”Deep learning models for predicting RNA degradation via dual crowdsourcing”的一项研究。作者巧妙地利用对两个众包平台的集成,获得能够对RNA降解进行极好预测的模型,以此来突破mRNA分子的热稳定性的限制。

研究背景和内容
基于mRNA的疗法显示出巨大的前景,快速部署针对COVID-19的基于mRNA的疫苗就证明了这一点。然而,mRNA分子在世界范围内的广泛使用受到RNA分子固有的不稳定性的限制。预测RNA分子的降解是设计更稳定的基于RNA的治疗方法的关键任务。
由于给定的治疗靶点可用的mRNA序列数量惊人,因此其中一些序列具有的结构特征,可能使其比第一代mRNA疫苗配方更耐水解。初步结果表明,可以通过优化候选RNA序列来设计用于模型蛋白质系统的更稳定的mRNA,并使用RNA水解模型进行评分。
然而,任何此类mRNA设计算法的潜力都受到基础模型预测RNA降解的准确性的限制。这就需要掌握在短时间内RNA降解可实现的最大预测能力,以便进行模型开发。为此,我们结合了两个众包平台:RNA设计平台Eterna和机器学习竞赛平台Kaggle。
我们使用来自Eterna平台上设计的短RNA片段的降解数据,该片段包含多种多样的序列和结构,并假设众包获得机器学习架构的问题将获得一个能够表达序列和结构依赖性降解模式的复杂性的模型(图1a)。我们假设这种“双重众包”将导致对开发的模型进行严格和独立的测试,最大限度地减少设计测试结构者和构建模型者之间的假设共享,并得到更好的独立数据集的泛化性。
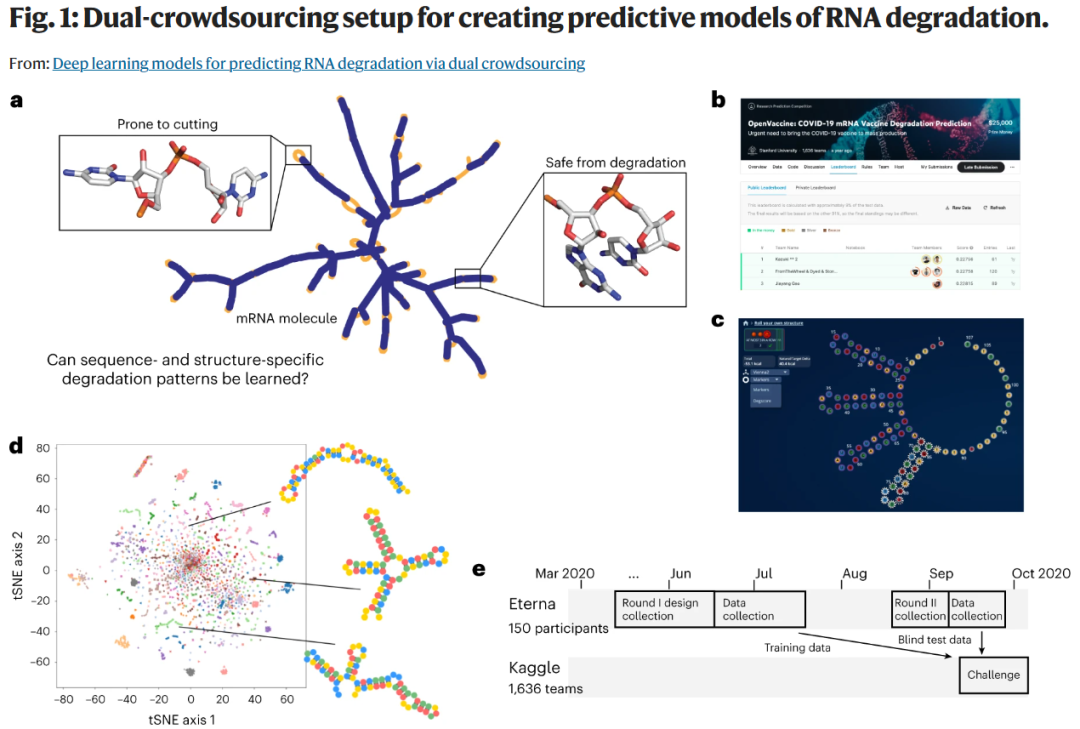
所得模型经受了两个盲测挑战。第一次是在Kaggle竞赛的背景下,参与者旨在预测的RNA结构探测和降解数据直到比赛宣布后才获得。在第二个盲测挑战中测试所得模型,预测编码多种模型蛋白的全长mRNA的整体降解。在预测这些总体降解率方面,这些模型的预测能力比现有方法更强。
研究结果
双众包竞赛设计与评估
OpenVaccine Kaggle竞赛(图1b)的目的是开发用于预测RNA降解模式的计算模型。我们要求Eterna平台上的参与者使用Web浏览器窗口提交RNA设计(图1c),这保证了序列和结构的多样性(图1d)。在第一轮比赛(RYOS-I)中总共收集了3029个长度为107 nt的RNA设计。
我们从 RYOS-I 数据集分出了训练和公共测试数据集如下图所示。公共测试数据集用于比赛期间对提交的内容进行排名。对3029个结构进行了平均信噪比值大于1的结构的过滤,得到2218个结构。将这些结构中1179个结构用作公共训练数据集、400个结构用作公共测试集和639个结构的私有测试数据集,其中公共测试集和私有测试集将在最终评估中使用。

为了确保用于私有测试集的大部分数据都是完全盲测的,我们发起了第二次挑战(RYOS-II)。RYOS-II实验与Kaggle挑战赛同时进行,从而对Kaggle上开发的模型进行了完全盲测。
Kaggle团队的表现和顶级模型的共同属性
在为期3周的比赛期间,1636支队伍提交了35806个解决方案。Kaggle条目在RNA降解方面明显优于“DegScore”线性回归模型,在MCRMSE中,公共测试集减少了37%,私有测试集减少了25%(下图a)。Kaggle参与者开发了超出所提供的特征编码,使用最广泛的社区开发的特征之一是图3b所示的基于图的距离嵌入。

我们探讨了是否可以通过集成模型来提高建模的准确性,即组合来自多个模型的预测。Kaggle竞赛的一个共同特点是不同的获胜的解决方案有很大不同,则将其集成模型经常提高预测能力。然而实验中集成模型只得到了适度的改进,这表明大部分信息已被前两个模型捕获。
顶级模型能够深度表示RNA基序
我们对比赛排名第一的模型(Nullrecurrent)的预测进行了更深入的分析,以更好地理解其性能。在私人测试集中的所有核苷酸中,41%的SHAPE反应性核苷酸水平预测与实验测量结果一致,误差幅度低于实验不确定性。相比之下,如果实验误差分布为正态分布,则完美预测变量将与多于68%的数据点的实验值一致。对于Deg_Mg_pH10和Deg_Mg_50C,分别有28%和42%的预测在误差范围内。
Kaggle模型改进了对mRNA降解的预测
作为一项独立测试,我们评估了前两个Kaggle模型预测全长mRNA数据集总体降解率的能力。由Eterna参与者设计的两种示例mRNA的实验确定的结构如下图b所示。两者都编码纳米荧光素酶,但在水解寿命上相差2.5倍。“Yellowstone”是由Eterna参与者设计的,使用密码子模仿黄石温泉中生物体的核苷酸频率,“LinearDesign-1”由Eterna参与者使用来自线性设计mRNA结构优化服务器的初始序列设计。
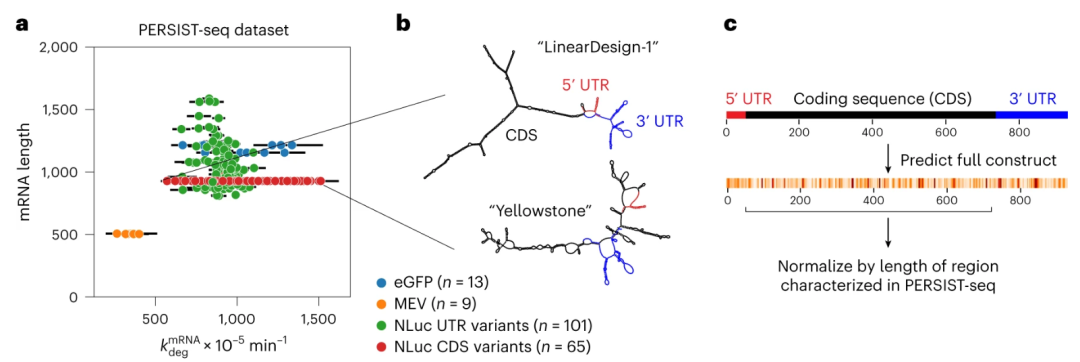
将Kaggle预测因子与PERSIST-seq的单个总降解速率进行比较,我们发现Kaggle排名第二的 “Kazuki2”模型与实验确定的降解率的相关性最高,其次是Kaggle排名第一的 “Nullrecurrent”模型。两个Kaggle模型的表现都优于ViennaRNA RNAfold v. 2.4.14、DegScore 线性回归模型和DegScore-XGBoost模型。
总结
OpenVaccine竞赛独特地利用了Kaggle和Eterna两个互补的众包平台的资源。在这场比赛中,大多数顶级团队在私人排行榜上的排名与他们在公共排行榜上的排名接近相同。因此该结果表明这些模型是稳健且可推广的,并且使用单独的、独立收集的数据集进行私人排行榜测试,对于确保可推广性非常重要。
这项工作中预测RNA降解的模型可以在随机mRNA设计算法中调用,以最大程度地减少可预测的降解,同时可能将被用于计算识别已进化为抗降解的天然RNA类别。这种未来的生物信息学分析可能为设计耐水解RNA疗法提供全新的生物学启发方法。
参考资料
Wayment-Steele, H.K., Kladwang, W., Watkins, A.M. et al. Deep learning models for predicting RNA degradation via dual crowdsourcing. Nat Mach Intell (2022).
https://doi.org/10.1038/s42256-022-00571-8


